Salud Intestinal, Bienestar y Más: El Papel Esencial de la Serotonina en el Organismo
La serotonina, conocida popularmente como la “hormona de la felicidad“,…
Sigue leyendo

Actualmente, el número de personas que presentan molestias relacionadas con el sistema digestivo está en aumento. En la mayoría de los casos, aunque no presenten síntomas graves, esto afecta la calidad de vida. El microbioma intestinal, con todos sus componentes e interacciones, forma un nicho ecológico completo que interactúa con nuestra fisiología.
El estilo de vida actual, con altos niveles de estrés, alimentación inadecuada o ciertos tratamientos, puede alterar el microbioma intestinal, causando un desequilibrio en los diversos tipos de microorganismos que habitan nuestro intestino, lo que puede tener consecuencias negativas para nuestra salud. Por lo tanto, conseguir un microbioma equilibrado es esencial para una buena salud (1).
El término microbioma se refiere al genoma colectivo de una comunidad microbiana, es decir, al conjunto completo de genes de los microorganismos que forman la microbiota, que incluye el material genético de todas las bacterias intestinales, virus, hongos y demás microorganismos.
Además, el término también se refiere a las actividades y capacidades metabólicas de los microorganismos presentes (2), como, por ejemplo, cuáles son los metabolitos producidos y consumidos por estos microorganismos.
La microbiota intestinal representa un ecosistema complejo que se desarrolla en paralelo con los hospedadores y depende del ambiente fisiológico de los mismos. La población bacteriana del intestino se estabiliza durante los primeros años de vida y permanece estable a lo largo de toda nuestra vida, estando dominada por cuatro filos principales: Firmicutes, Bacteroidetes, Actinobacteria y Proteobacteria.
Se estima que el número de especies de microorganismos en el intestino humano es de entre 1,000 y 1,150, y el número de genes del microbioma intestinal excede el número de genes en el genoma humano en 150 veces (3).
La microbiota intestinal tiene la capacidad de llevar a cabo muchos procesos que no pueden ser ejecutados por el hospedero. Estos procesos pueden resultar en la producción o modulación de metabolitos microbianos que funcionan como sustratos metabólicos y moléculas señalizadoras en el hospedero, con importantes implicaciones para el metabolismo y la salud del mismo.
La composición de la dieta es fundamental para la producción metabólica de la microbiota intestinal, ya que procesa los nutrientes dietéticos en metabolitos. Por lo tanto, la dieta afecta la composición de la microbiota intestinal y, así, su potencial metabólico e impacto en el hospedeiro (4).
La microbiota adquirida al nacer se desarrolla en paralelo con el desarrollo del hospedero y mantiene su estabilidad temporal y diversidad a lo largo de la vida adulta hasta la muerte.
Los estudios de la literatura sugieren que, aunque una parte de la microbiota se conserva, los miembros dinámicos varían a lo largo del tracto gastrointestinal, desde bebés hasta ancianos, en diferentes condiciones de salud.
Aunque la microbiota intestinal es dinámica, desempeña algunas funciones básicas en los escenarios inmunológico, metabólico, estructural y neurológico del cuerpo humano.
La microbiota intestinal también ejerce una influencia significativa tanto en la salud física como mental del individuo (5),
ya que regula muchos procesos metabólicos en el huésped, incluyendo la homeostasis energética, el metabolismo de la glucosa y el metabolismo lipídico (6).
La microbiota intestinal desempeña sus funciones en cuatro contextos distintos en el cuerpo humano (7):
Las complejas comunidades de microorganismos que habitan el tracto gastrointestinal humano están emergiendo como elementos clave en la regulación de la salud y la enfermedad. Varias funciones vitales desempeñadas por el microbioma intestinal en el organismo humano resaltan su importancia.
Esto incluye la transformación de partes no digeribles de los alimentos en sustancias que pueden ser absorbidas; la producción de vitaminas esenciales; la eliminación de sustancias tóxicas; la competencia con patógenos; el fortalecimiento de la barrera intestinal y la regulación del sistema inmunológico.
Muchas de estas funciones están estrechamente relacionadas con la fisiología humana. Por ejemplo, los ácidos grasos de cadena corta producidos por la fermentación microbiana son importantes para las células intestinales y desempeñan roles esenciales en la regulación del sistema inmunológico (8-10).
En las etapas iniciales de la vida, el desarrollo del microbioma influye en la función inmunológica. Los microorganismos adquiridos de manera vertical, horizontal y del ambiente, así como sus productos metabólicos, tienen el potencial de moldear los cursos de desarrollo que impactan la salud a lo largo de la vida (11).
Así, el microbioma desempeña un papel crucial en el desarrollo de funciones metabólicas, inmunológicas y nutricionales, lo que hace necesario un cuidado atento. De esta manera, comprender cómo las comunidades microbianas complejas pueden afectar la patogénesis de diversas enfermedades tiene implicaciones significativas para su prevención, diagnóstico y tratamiento (12).
En las últimas décadas, el campo de la investigación del microbioma humano ha evolucionado más allá de la simple catalogación de la diversidad de microorganismos, hacia la comprensión de cómo estos microorganismos constituyen un sistema funcional auxiliar y dinámico, que se desarrolla de manera sinérgica en paralelo con el proceso de desarrollo y declive fisiológico (13-15).
Lo que es cada vez más evidente es que una amplia gama de condiciones, que incluye enfermedades inflamatorias crónicas, está estrechamente relacionada con la composición y el equilibrio del microbioma intestinal (16), enfermedades metabólicas (17), trastornos neurológicos y cáncer. (18, 19), ahora están siendo relacionadas con alteraciones funcionales en el microbioma.
Estas alteraciones pueden ocurrir tanto de manera aislada en el lugar de manifestación de la enfermedad como en áreas de mucosas o sistemas de órganos distantes, lo que desencadena cambios metabólicos e inmunológicos en el huésped (20).
Diversos factores, como la dieta, los agentes antimicrobianos y la inmunidad, influyen en los microbiomas humanos, especialmente el microbioma intestinal, que alberga la mayor cantidad y variedad de microorganismos. En respuesta, los productos bioactivos provenientes del microbioma moldean la función de las células humanas (2, 21).
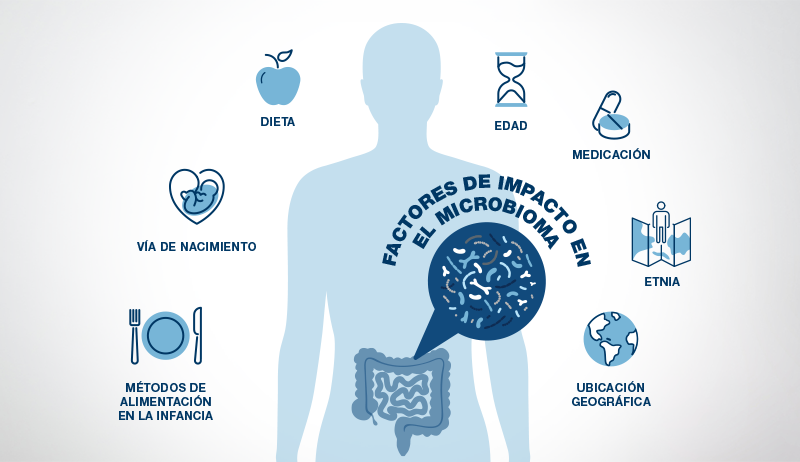
Después de cumplir dos o tres años de edad, el microbioma intestinal se vuelve más estable y, en esta etapa, se parece mucho al microbioma de un adulto (20). Sin embargo, el microbioma intestinal del adulto todavía puede verse influenciado por muchos factores. La dieta es la que más influye, seguida de los medicamentos.
El estrés también juega un papel importante, ya que el cerebro puede influir en el microbioma intestinal enviando hormonas del estrés al intestino a través del nervio vago. Los hábitos de ejercicio, la edad, la genética, la ubicación geográfica y la etnia también son elementos que afectan el microbioma intestinal (16).
Los desequilibrios en el microbioma intestinal pueden ser cualitativos o cuantitativos y afectar tanto su distribución como su actividad, lo que tiene un efecto comprobado sobre la salud intestinal y general del individuo.
‘Las enfermedades relacionadas con el desequilibrio del microbioma intestinal incluyen afecciones como:
El mecanismo de esta relación se debe al hecho de que los microorganismos pueden producir diferentes metabolitos biológicamente activos, y estos metabolitos pueden desempeñar un papel en un estado de enfermedad.
La causalidad entre el intestino y cada enfermedad aún está siendo establecida y no se sabe si la enfermedad causa alteraciones en el microbioma intestinal, o si un cambio en el microbioma intestinal causa la enfermedad.
Varias enfermedades intestinales relacionadas con la inflamación han sido correlacionadas con alteraciones en el microbioma intestinal. Esto incluye enfermedades como el cáncer de colon, la colitis ulcerosa, la enfermedad de Crohn y el síndrome del intestino irritable (3, 22, 23).
La enfermedad de Crohn (EC) es una enfermedad inflamatoria del intestino (EII) que puede afectar cualquier parte del tracto gastrointestinal. La EC se ha relacionado con anomalías funcionales que favorecen un fenotipo proinflamatorio marcado y desequilibrios estructurales en el ecosistema intestinal, lo que resulta en una disminución en las tasas de crecimiento de microorganismos beneficiosos y un aumento en las tasas de crecimiento de ciertas bacterias patógenas u oportunistas.
Los microorganismos intestinales han recibido mucha atención recientemente como posibles impulsores de la EC. Esta idea se ve corroborada por el hecho de que la estrecha interacción entre la microbiota intestinal y la mucosa intestinal influye continuamente en el sistema inmunológico intestinal, y cualquier desequilibrio en el estado normal del microbioma probablemente pueda desencadenar una disfunción inmunológica a través de señales proinflamatorias (24).
Con el avance de la tecnología metagenómica, evidencias crecientes sugieren que la disbiosis intestinal, es decir, el desequilibrio en la microbiota intestinal normal, puede promover condiciones inflamatorias crónicas y la producción de metabolitos carcinogénicos, lo que lleva a la neoplasia (25).
Aunque los genes contribuyen al cáncer colorrectal, la microbiota intestinal desempeña un papel importante. Evidencias científicas sugieren que la infección crónica y la inflamación subsiguiente contribuyen a la iniciación y progresión del tumor.
Se han investigado diversas especies bacterianas y mecanismos de virulencia promotores de tumores. Se han logrado avances significativos en la comprensión de la composición y capacidades funcionales del microbioma intestinal y su papel en el câncer (3).
Se ha demostrado que algunas bacterias aumentan consistentemente (Fusobacteria, Alistipes, Porphyromonadaceae, Coriobacteridae, Staphylococcaceae, Akkermansia spp., Methanobacteriales), mientras que otras disminuyen constantemente en el cáncer colorrectal (Bifidobacterium, Lactobacillus, Ruminococcus, Faecalibacterium spp., Roseburia, Treponema). También se ha evidenciado que los metabolitos bacterianos de aminoácidos están aumentados y que el butirato está disminuido a lo largo de la carcinogénesis. (26).
La enfermedad celíaca es un trastorno digestivo que afecta aproximadamente al 1% de la población, aunque un porcentaje mucho mayor porta genes relacionados con la susceptibilidad genética.
Un estudio demostró que el microbioma podría ser un cofactor en el desarrollo de la enfermedad en personas genéticamente predispuestas. El estudio, realizado en ratones, mostró que el microbioma duodenal puede proteger o provocar la enfermedad celíaca en ratones genéticamente susceptibles, dependiendo de la composición microbiana y del equilibrio entre patobiontes.
Es decir, los microorganismos que tienen potencial para causar enfermedades pueden hacerlo si las condiciones son favorables, mientras que los microorganismos comensales, que viven en simbiosis con el hospedero y se benefician de la relación sin causar daños (27).
La depresión es uno de los trastornos mentales más comunes. Sin embargo, sus causas aún son parcialmente desconocidas y su diagnóstico es complejo. Un grupo de investigadores holandeses detectó variaciones en el microbioma intestinal que son características de individuos con depresión y verificaron que tales variaciones son independientes del grupo étnico.
El estudio señala que algunas de las bacterias involucradas son productoras de metabolitos como el glutamato, el butirato, la serotonina y el ácido gamma-aminobutírico (GABA), que son sustancias con potente acción neurológica. Estas sustancias ejercerían su efecto a través del nervio vago, que conecta el intestino y el cerebro, influyendo en el estado de ánimo y en la depresión (28).
Con los avances tecnológicos, el conocimiento sobre el microbioma intestinal ha experimentado un considerable crecimiento en los últimos años. La influencia del microbioma intestinal en la salud y la enfermedad es actualmente una de las áreas más exploradas de la ciencia.
Profundizar nuestra comprensión sobre cómo los trastornos del microbioma intestinal en las primeras etapas de la vida, combinados con otros factores estresantes a lo largo de la vida, afectan la salud, puede proporcionar nuevas ideas para el tratamiento de muchos trastornos y enfermedades complejas (como la fibromialgia, enfermedad inflamatoria intestinal, autismo).
Las investigaciones futuras deben enfocarse en el microbioma intestinal como biomarcador para la ingesta alimentaria y el desarrollo de enfermedades, el efecto de la suplementación probiótica en los resultados clínicos y la terapia bacteriana para la salud intestinal, la inmunidad y los tratamientos terapêuticos (29).

SYNLAB ofrece el examen MyBiome, que es la única prueba diagnóstica del microbioma intestinal, ya que realiza la lectura completa del genoma del microorganismo intestinal mediante secuenciación masiva (shotgun metagenomics), lo que permite llevar a cabo un estudio exhaustivo, objetivo y práctico de todos los microorganismos que componen el ecosistema intestinal, proporcionando al análisis:
De esta manera, el MyBiome proporciona información detallada sobre los microorganismos que habitan el intestino y su funcionalidad, el impacto en la salud, qué funciones clave para la salud están siendo realizadas por los microorganismos detectados y cómo lograr un equilibrio a través de recomendaciones nutricionales personalizadas.
El examen MyBiome se realiza mediante secuenciación shotgun, una técnica metagenómica que consiste en la fragmentación aleatoria de fragmentos de ADN, seguido por el secuenciamiento individual de estos fragmentos.
Este enfoque es altamente efectivo para secuenciar genomas completos, especialmente de organismos cuyo genoma es demasiado grande para ser secuenciado de forma convencional. De esta manera, la secuenciación shotgun permite un análisis completo del genoma de todas las bacterias y microorganismos presentes en la microbiota intestinal.
A través de la secuenciación metagenómica, es posible obtener información exhaustiva sobre el genoma de un organismo, lo que permite el análisis de genes, vías metabólicas y regiones reguladoras, ya que se secuencia todo el genoma, que incluye millones a miles de millones de pares de bases.
En contraste, la secuenciación del gen 16S se emplea solo para identificar y clasificar las bacterias presentes en una muestra, ya que solo se secuencia una región específica del gen 16S rRNA, relativamente corta (aproximadamente 1,500 bases) (30).
El uso de secuenciación metagenómica (shotgun) para el análisis de la microbiota intestinal presenta diversas ventajas en comparación con los estudios basados en la secuenciación del gen 16S o PCR-RT.
El análisis convencional de poblaciones bacterianas mediante la amplificación del gen del ARN ribosomal 16S (16S rRNA) se basa en la amplificación de regiones hipervariables de este gen mediante cebadores (secuencias iniciadoras de bases que se complementan y se unen específicamente con las regiones de interés en el ADN), que amplifican pequeñas regiones del 16S en la mayoría de las bacterias presentes en una muestra.
Sin embargo, esta técnica no permite detectar microorganismos que hayan experimentado modificaciones en el sitio de unión de los cebadores (31). Por lo tanto, estas bacterias son técnicamente “invisibles” y escapan al análisis (32).
Además, el gen 16S rRNA representa solo una pequeña parte de todo el genoma de una bacteria, lo que dificulta cualquier tipo de análisis más allá de la clasificación filogenética.
En comparación con los estudios convencionales de la microbiota basados en el análisis del 16S rRNA, MyBiome permite detectar todos los genes presentes en los microorganismos de la muestra gracias al uso de la técnica de secuenciación metagenómica, evitando sesgos de amplificación y facilitando un muestreo completo de todos los genes presentes en los organismos (33).
El enfoque metagenómico proporciona un análisis más profundo, brindando información no solo sobre todas las bacterias presentes hasta el nivel taxonómico de especie y cepa, sino también sobre otros microorganismos presentes en la muestra, como hongos, arqueas y protistas.
Además de la identificación, la secuenciación metagenómica permite la caracterización y cuantificación de genes funcionales, proporcionando información sobre las funciones de los microorganismos presentes en la muestra.
La tecnología utilizada en MyBiome también se destaca por su alta resolución en comparación con el análisis convencional de amplificación del gen 16S rRNA. Mientras que la secuenciación del gen 16S no permite discriminar entre especies bacterianas con regiones 16S similares o idénticas (34), El secuenciamento metagenómico permite una identificación más precisa de los microorganismos y sus genes funcionales.
Esta limitación implica una pérdida de información significativa, ya que diferentes especies de un mismo género pueden tener funciones muy distintas (35). La secuenciación masiva de todo el genoma utilizada en MyBiome es actualmente la técnica de mayor resolución para identificar microorganismos y detectar sus genes funcionales.
Estas características, junto con recomendaciones personalizadas, hacen que MyBiome sea un análisis único en el mercado, proporcionando información detallada sobre la microbiota intestinal y su funcionalidad.
El MyBiome está especialmente indicado para:
La realización de exámenes precisos y actualizados, como el MyBiome, es esencial para realizar diagnósticos más precisos y dirigir mejor los tratamientos. Y SYNLAB está aquí para ayudarte.
Ofrecemos soluciones diagnósticas con un estricto control de calidad a empresas, pacientes y médicos a los que atendemos. Llevamos más de 10 años en Brasil, operamos en 36 países y tres continentes, y somos líderes en la prestación de servicios en Europa.
¡ Entre en contacto con el equipo de SYNLAB y conoce los exámenes disponibles!
Referencias Bibliográficas
(1) Berg G, et al. Microbiome definition re-visited: old concepts and new challenges. Microbiome. 2020 Jun 30;8(1):103. doi: 10.1186/s40168-020-00875-0.
(2) Human Microbiome Project Consortium. Structure, function and diversity of the healthy human microbiome. Nature. 2012;486(7402), 207-214.
(3) Sun J; Kato I. Gut microbiota, inflammation and colorectal câncer. Genes Dis. 2016 Jun;3(2):130-143.
(4) Schoeler Marc; Caesar Robert. Dietary lipids, gut microbiota and lipid metabolismo. Reviews in Endocrine and Metabolic Disorders (2019) 20:461–472.
(5) Atanu Adak; Mojibur R. Khan. An insight into gut microbiota and its functionalities. Cellular and Molecular Life Sciences (2019) 76:473–493.
(6) Sonnenburg JL, Backhed F. Diet-microbiota interactions as moderators of human metabolism. Nature. 2016;535(7610):56–64.
(7) Sarkar A, Lehto SM, Harty S, Dinan TG, Cryan JF, Burnet PW. (2016) Psychobiotics and the manipulation of bacteria–gut–brain signals. Trends Neurosci 39(11):763–781.
(8) LeBlanc, J.G. et al. (2013) Bacteria as vitamin suppliers to their host: a gut microbiota perspective. Curr. Opin.Biotechnol. 24, 160–168
(9) Claus, S.P. et al. (2016) The gut microbiota: a major player in the toxicity of environmental pollutants? NPJ Biofilms Microbiomes 2, 16003
(10) Kamada, N. et al. (2013) Role of the gut microbiota inimmunity and inflammatory disease. Nat. Rev.Immunol. 13, 321–335).
(11) Ursell LK, Metcalf JL, Parfrey LW, Knight R. Defning the human microbiome. Nutr Ver. 2012;70(Suppl 1):S38-44.
(12) Hollister, E. B. et al. Structure and function of the healthy pre-adolescent pediatric gut microbiome. Microbiome. 2015;3,36.
(13) Claesson, M. J. et al. Gut microbiota composition correlates with diet and health in the elderly. Nature. 2012;488,178–184.
(14) Stewart, C. J. et al. Temporal development of the gut microbiome in early childhood from the TEDDY study. Nature. 2018;562,583–588.
(15) Arrieta MC, Stiemsma LT, Amenyogbe N, Brown EM, Finlay B. The intestinal microbiome in early life: health and disease. Front. Immunol. 2014;5,427.
(16) Le Chatelier, E. et al. Richness of human gut microbiome correlates with metabolic markers. Nature. 2013;500,541–546.
(17) Cryan, J. F., O’Riordan, K. J., Sandhu, K., Peterson, V. & Dinan, T. G. The gut microbiome in neurological disorders. Lancet Neurol. 2020;19,179–194.
(18) Nejman, D. et al. The human tumor microbiome is composed of tumor type specific intracellular bacteria. Science. 2020;368,973–980.
(19) Hepworth, M. R. et al. Innate lymphoid cells regulate CD4+ T-cell responses to intestinal commensal bacteria. Nature. 2013;498, 113–117.
(20) Rackaityte E, Lynch SV. The human microbiome in the 21st century. Nat Commun. 2020 Oct 16;11(1):5256. doi: 10.1038/s41467-020-18983-8.
(21) Scharschmidt, T. C. et al. A wave of regulatory T Cells into neonatal skin mediates tolerance to commensal microbes. Immunity. 2015;43,1011–1021.
(22) Qing He, Yuan Gao, Zhuye Jie, Xinlei Yu, Janne Marie Laursen. Two distinct metacommunities characterize the gut microbiota in Crohn’s disease patients. Gigascience. 2017 Jul 1;6(7):1-11.
(23) Katsuyoshi Matsuoka 1, Takanori Kanai. The gut microbiota and inflammatory bowel disease. Semin Immunopathol. 2015 Jan;37(1):47-55.
(24) He Q, et al. Two distinct metacommunities characterize the intestinal microbiota in patients with Crohn’s disease. Gigascience. 2017 Jul 1;6(7):1-11.
(25) Marchesi JR, Dutilh BE, Hall N, et al. Towards the human colorectal cancer microbiome. PLoS One. 2011;6:e20447.
(26) Borges-Canha M, et al. Role of colonic microbiota in colorectal carcinogenesis: a systematic review. Rev Esp Enferm Dig. 2015;107.
(27) Constante M, et al. Biogeographic Variation and functional pathways of the Gut microbiota in Celiac Disease. Gastroenterology 2022 163(5):1351-1363.e15.
(28) Radjabzadeh D, eta l. Gut microbiome-wide association study of depressive symptoms. Nat Commun. 2022. 6;13(1)7128.
(29) Cresci G, Bawden E. The Gut Microbiome: What we do and don’t know. Nutr Clin Pract. 2015 December; 30(6): 734–746.
(30) Ranjan R, Rani A, Metwally A, McGee HS, Perkins DL. Analysis of the microbiome: Advantages of whole genome shotgun versus 16S amplicon sequencing. Biochem Biophys Res Commun. 2016;22;469(4):967-77.
(31) Yang B, Wang Y, Qian PY. Sensitivity and correlation of hypervariable regions in 16S rRNA genes in phylogenetic analysis. BMC Bioinformatics. 2016 Mar 22;17:135.
(32) Větrovský T, Baldrian P. The variability of the 16S rRNA gene in bacterial genomes and its consequences for bacterial community analyses. PLoS One. 2013;8(2):e57923.
(33) Kumar S, Krishnani KK, Bhushan B, Brahmane MP. Metagenomics: Retrospect and Prospects in High Throughput Age. Biotechnol Res Int. 2015;2015:121735.
(34) Fox GE, Wisotzkey JD, Jurtshuk P Jr. How close is close: 16S rRNA sequence identity may not be sufficient to guarantee species identity. Int J Syst Bacteriol.1992 Jan;42(1):166-70.
(35) Clarridge JE 3rd. Impact of 16S rRNA gene sequence analysis for identification of bacteria on clinical microbiology and infectious diseases. Clin Microbiol Rev. 2004 Oct;17(4):840-62.
Infórmate sobre el examen
La serotonina, conocida popularmente como la “hormona de la felicidad“,…
Sigue leyendo
¿Alguna vez has sentido malestar después de comer ciertos alimentos,…
Sigue leyendo
La dermatomiositis es una enfermedad inflamatoria rara que afecta la…
Sigue leyendo