Doenças hepáticas: impacto, prevenção e diagnóstico precoce
Doenças hepáticas representam uma carga significativa para a saúde global, com…
Continuar lendo

Atualmente, é crescente o número de pessoas que apresentam desconfortos relacionados ao sistema digestivo. Na maioria dos casos, mesmo sem sintomas graves, isso afeta a qualidade de vida. O microbioma intestinal, com todos seus componentes e interações, forma um nicho ecológico completo que interage com nossa fisiologia.
O estilo de vida atual, com altos níveis de estresse, alimentação inadequada ou certos tratamentos, pode alterar o microbioma intestinal, causando um desequilíbrio nos diversos tipos de microrganismos que habitam nosso intestino, o que pode ter consequências negativas para a nossa saúde. Dessa forma, conseguir um microbioma equilibrado é essencial para uma boa saúde (1).
O termo microbioma refere-se ao genoma coletivo de uma comunidade microbiana, ou seja, é o conjunto completo de genes dos microrganismos que formam a microbiota, que inclui o material genético de todas as bactérias intestinais, vírus, fungos e demais microrganismos.
Além disso, o termo refere-se ainda sobre as atividades e capacidades metabólicas dos microrganismos presentes (2), como, por exemplo, quais os metabólitos produzidos e consumidos por esses microrganismos.
A microbiota intestinal representa um ecossistema complexo que se desenvolve em paralelo com os hospedeiros e depende do ambiente fisiológico dos mesmos. A população bacteriana do intestino se estabiliza durante os primeiros anos de vida e permanece estável ao longo de toda a nossa vida, sendo dominada por quatro principais filos: Firmicutes, Bacteroidetes, Actinobacteria e Proteobacteria.
O número de espécies de microrganismos no intestino humano é estimado em 1.000 a 1.150, sendo que o número de genes do microbioma intestinal excede o número de genes no genoma humano em 150 vezes (3).
A microbiota intestinal tem a capacidade de realizar muitos processos que não podem ser executados pelo hospedeiro. Esses processos podem resultar em metabólitos produzidos ou modulados microbianamente que funcionam como substratos metabólicos e moléculas sinalizadoras no hospedeiro, com importantes implicações para o metabolismo e a saúde do mesmo.
A composição dietética é fundamental para a produção metabólica da microbiota intestinal, uma vez que processa nutrientes dietéticos em metabólitos, desta forma, a dieta afeta a composição da microbiota intestinal e, assim, seu potencial metabólico e impacto no hospedeiro (4).
A microbiota adquirida no nascimento se desenvolve em paralelo com o desenvolvimento do hospedeiro e mantém sua estabilidade temporal e diversidade ao longo da vida adulta até a morte.
Os estudos da literatura sugerem que, embora uma parte da microbiota seja conservada, membros dinâmicos variam ao longo do trato gastrointestinal, desde bebês até idosos, em diferentes condições de saúde.
Embora a microbiota intestinal seja dinâmica, ela desempenha algumas funções básicas nos cenários imunológico, metabólico, estrutural e neurológico do corpo humano.
A microbiota intestinal também exerce uma influência significativa tanto na saúde física quanto mental do indivíduo (5), uma vez que regula muitos processos metabólicos no hospedeiro, incluindo homeostase energética, metabolismo da glicose e metabolismo lipídico (6).
A microbiota intestinal desempenha suas funções em quatro contextos distintos no corpo humano (7):
As complexas comunidades de microorganismos que habitam o trato gastrointestinal humano estão se destacando como elementos-chave na regulação da saúde e doença. Diversas funções vitais desempenhadas pelo microbioma intestinal no organismo humano destacam sua importância.
Isso inclui a transformação de partes não digeríveis dos alimentos em substâncias que podem ser absorvidas; produção de vitaminas essenciais; eliminação de substâncias tóxicas; competição com patógenos; fortalecimento da barreira intestinal e regulação do sistema imunológico.
Muitas dessas funções estão intimamente ligadas à fisiologia humana. Por exemplo, os ácidos graxos de cadeia curta produzidos pela fermentação microbiana são importantes para as células intestinais e desempenham papéis essenciais na regulação do sistema imunológico (8-10).
Nos estágios iniciais da vida, o desenvolvimento do microbioma influencia a função imunológica. Microrganismos adquiridos verticalmente, horizontalmente e do ambiente, bem como seus produtos metabólicos, têm o potencial de moldar os cursos de desenvolvimento que impactam a saúde ao longo da vida (11).
Assim, o microbioma desempenha um papel crucial no desenvolvimento de funções metabólicas, imunológicas e nutricionais, tornando necessário um cuidado atento. Desta forma, compreender como comunidades microbianas complexas podem afetar a patogênese de diversas doenças tem implicações significativas para prevenção, diagnóstico e tratamento das mesmas (12).
Nas últimas décadas, o campo da pesquisa do microbioma humano evoluiu além da simples catalogação da diversidade de microrganismos, para a forma como esses microrganismos constituem um sistema funcional auxiliar e dinâmico, que se desenvolve de maneira sinérgica, em paralelo com o processo de desenvolvimento e declínio fisiológico (13-15).
O que está cada vez mais evidente é que uma ampla gama de condições, que inclui doenças inflamatórias crônicas (16), doenças metabólicas (17), distúrbios neurológicos e câncer (18, 19), agora está sendo relacionado a alterações funcionais no microbioma.
Essas alterações podem ocorrer tanto isoladamente no local de manifestação da doença quanto em áreas de mucosas ou sistemas de órgãos distantes, o que desencadeia mudanças metabólicas e imunológicas no hospedeiro (20).
Diversos fatores, como a dieta, agentes antimicrobianos e imunidade, influenciam os microbiomas humanos, especialmente o microbioma intestinal, que abriga a maior quantidade e variedade de microrganismos. Em resposta, os produtos bioativos proveniente do microbioma moldam a função das células humanas (2, 21).
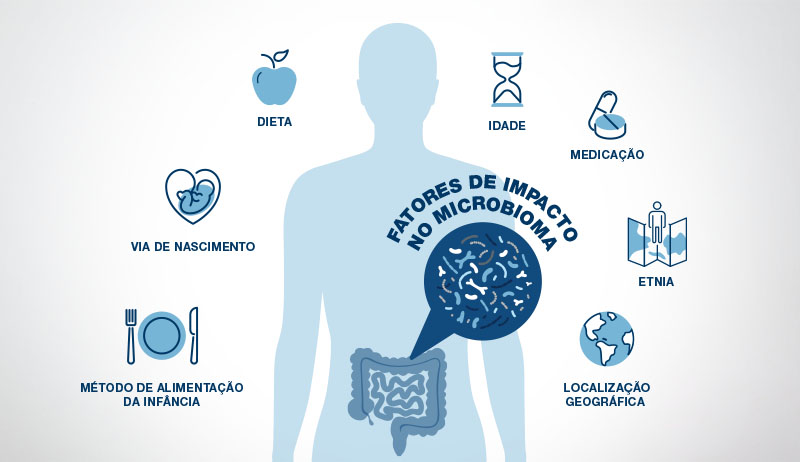
Após atingirmos entre dois e três anos de idade, o microbioma intestinal se torna mais estável e, nessa fase, se assemelha muito ao microbioma de um adulto (20). No entanto, o microbioma intestinal adulto ainda pode ser influenciado por muitos fatores. A dieta é o mais influente deles, seguido pelos medicamentos.
O estresse também desempenha um papel importante, pois o cérebro pode influenciar o microbioma intestinal enviando hormônios do estresse para o intestino através do nervo vago. Hábitos de exercício, idade, genética, localização geográfica e etnia também são elementos que atuam no microbioma intestinal (16).
Os desequilíbrios no microbioma intestinal podem ser qualitativos ou quantitativos e afetar tanto sua distribuição quanto sua atividade, o que tem um efeito comprovado sobre a saúde intestinal e geral do indivíduo.
As doenças relacionadas ao desequilíbrio do microbioma intestinal incluem condições como:
O mecanismo dessa relação decorre do fato de que os microrganismos podem produzir diferentes metabólitos biologicamente ativos, e esses metabólitos podem desempenhar um papel em um estado de doença.
A causalidade entre o intestino e cada doença ainda está sendo estabelecida e não se sabe se a doença causa alterações no microbioma intestinal, ou se uma mudança no microbioma intestinal causa a doença.
Várias doenças intestinais ligadas à inflamação foram correlacionadas com alterações no microbioma intestinal. Isso inclui doenças como câncer de cólon, colite ulcerativa, doença de Crohn e síndrome do intestino irritável (3, 22, 23).
A doença de Crohn (DC) é uma doença inflamatória do intestino (DII) que pode afetar qualquer parte do trato gastrointestinal. A DC foi relacionada a anomalias funcionais que favorecem um fenótipo pró-inflamatório marcante e a desequilíbrios estruturais no ecossistema intestinal, resultando em uma diminuição nas taxas de crescimento de microrganismos benéficos e um aumento nas taxas de crescimento de certas bactérias patogênicas ou oportunistas.
Os microrganismos intestinais recentemente ganharam muita atenção como possíveis impulsionadores da DC. Essa ideia corroborada pelo fato de que a estreita interação entre a microbiota intestinal e a mucosa do intestino influencia continuamente o sistema imunológico intestinal, e qualquer desequilíbrio do estado normal do microbioma, pode provavelmente desencadear uma disfunção imunológica através de sinais pró-inflamatórios (24).
Com o avanço da tecnologia metagenômica, evidências crescentes sugerem que a disbiose intestinal, ou seja, o desequilíbrio na microbiota intestinal normal, pode promover condições inflamatórias crônicas e a produção de metabólitos carcinogênicos, levando à neoplasia (25).
Embora os genes contribuam para o câncer colorretal, a microbiota intestinal desempenha um papel importante. Evidências científicas sugerem que a infecção crônica e a subsequente inflamação contribuem para a iniciação e progressão do tumor.
Diversas espécies bacterianas e mecanismos de virulência promotores de tumores têm sido investigados. Avanços significativos foram feitos na compreensão da composição e capacidades funcionais do microbioma intestinal e seu papel no câncer (3).
Foi demonstrado que algumas bactérias são consistentemente aumentadas (Fusobacteria, Alistipes, Porphyromonadaceae, Coriobacteridae, Staphylococcaceae, Akkermansia spp., Methanobacteriales), enquanto outras são constantemente diminuídas no câncer colorretal (Bifidobacterium, Lactobacillus, Ruminococcus, Faecalibacterium spp., Roseburia, Treponema). Também evidenciou-se que metabólitos bacterianos de aminoácidos estão aumentados e o butirato está diminuído ao longo da carcinogênese (26).
A doença celíaca é um distúrbio digestivo que afeta aproximadamente 1% da população, embora uma porcentagem muito maior seja portadora de genes relacionados à susceptibilidade genética.
Um estudo demonstrou que o microbioma poderia ser um cofator no desenvolvimento da doença em pessoas geneticamente predispostas. O estudo, realizado em camundongos, mostrou que o microbioma duodenal pode proteger ou provocar a doença celíaca em camundongos geneticamente susceptíveis, dependendo da composição microbiana e do equilíbrio entre patobiontes.
Ou seja, microrganismos que têm potencial para causar doenças se as condições forem favoráveis e comensais, que são os microrganismos que vivem em simbiose com o hospedeiro, beneficiando-se da relação sem causar danos (27).
A depressão é um dos distúrbios mentais mais comuns. No entanto, suas causas ainda são parcialmente desconhecidas e seu diagnóstico é complexo. Um grupo de pesquisadores holandeses detectou variações no microbioma intestinal que são características de indivíduos com depressão e verificaram que tais variações são independentes do grupo étnico.
O estudo aponta que algumas das bactérias envolvidas são produtoras de metabólitos como glutamato, butirato, serotonina e ácido gama-aminobutírico (GABA), que são substâncias com potente ação neurológica. Essas substâncias exerceriam seu efeito através do nervo vago, que conecta o intestino e o cérebro, influenciando no estado de ânimo e na depressão (28).
Com os avanços tecnológicos, o conhecimento sobre o microbioma intestinal expandiu-se de forma considerável nos últimos anos. A influência do microbioma intestinal na saúde e na doença é atualmente uma das áreas mais exploradas da ciência.
Aprofundar nossa compreensão sobre como distúrbios ao microbioma intestinal no início da vida, combinados com outros estresses ao longo da vida, impactam a saúde, pode fornecer novas ideias para o tratamento de muitos distúrbios e doenças complexas (como a fibromialgia, doença inflamatória intestinal, autismo).
Pesquisas futuras devem direcionar o foco para o microbioma intestinal como biomarcador para consumo alimentar e desenvolvimento de doenças, o efeito da suplementação probiótica em desfechos clínicos e terapia bacteriana para saúde intestinal, imunidade e tratamentos terapêuticos (29).

A SYNLAB oferece o exame MyBiome, único teste diagnóstico do microbioma intestinal, pois realiza a leitura completa do genoma do microbiano intestinal por meio de sequenciamento massivo (shotgun metagenomics), o que permite realizar um estudo aprofundado, objetivo e acionável de todos os microrganismos que compõem o ecossistema intestinal, proporcionando à análise:
Desta forma, o MyBiome fornece informações detalhadas sobre os microrganismos que habitam o intestino e sua funcionalidade, o impacto na saúde, quais funções chave para a saúde estão sendo realizas pelos microrganismos detectados e como alcançar um equilíbrio através de recomendações nutricionais personalizadas.
O exame MyBiome é realizado mediante sequenciamento shotgun, uma técnica metagenômica que consiste na fragmentação aleatória de fragmentos de DNA, seguido pelo sequenciamento individual desses fragmentos.
Essa abordagem é altamente eficaz para sequenciar genomas inteiros, especialmente de organismos cujo genoma é grande demais para ser sequenciado de forma convencional. Desta forma, o sequenciamento shotgun possibilita uma análise completa do genoma de todas as bactérias e microrganismos presentes na microbiota intestinal.
Por meio do sequenciamento metagenômico, é possível obter informações abrangentes sobre o genoma de um organismo, permitindo a análise de genes, vias metabólicas e regiões regulatórias, uma vez que o genoma todo é sequenciado, o que inclui milhões a bilhões de pares de bases.
Em contraste, o sequenciamento do gene 16S é empregado apenas para identificar e classificar bactérias presentes em uma amostra, já que apenas uma região específica do gene 16S rRNA, relativamente curta (cerca de 1.500 bases), é sequenciada (30).
O uso do sequenciamento metagenômico (shotgun) para a análise da microbiota intestinal apresenta diversas vantagens em relação aos estudos baseados no sequenciamento do gene 16S ou PCR-RT.
A análise convencional de populações bacterianas por amplificação do gene RNA ribossomal 16s (16S RNAr) se baseia na amplificação de regiões hipervariáveis deste gene por meio de primers (sequências iniciadoras de bases que se complementam e se ligam especificamente com as regiões de interesse no DNA), que amplificam pequenas regiões do 16S na maioria das bactérias presentes em uma amostra.
No entanto, esta técnica não permite detectar microrganismos que sofreram modificações no sítio de ligação dos primers (31). Portanto, essas bactérias são tecnicamente “invisíveis” e escapam à análise (32).
Ademais, o gene 16S rRNA representa apenas uma pequena parte de todo o genoma de uma bactéria, o que dificulta qualquer tipo de análise além da classificação filogenética.
Comparado aos estudos convencionais da microbiota baseados na análise do 16S rRNA, o MyBiome permite detectar todos os genes presentes nos microrganismos da amostra graças ao uso da técnica de sequenciamento metagenômico, evitando viés de amplificação e facilitando uma amostragem completa de todos os genes presentes nos organismos (33).
A abordagem metagenômica proporciona uma análise mais profunda, fornecendo informações não apenas sobre todas as bactérias presentes até o nível taxonômico de espécie e cepa, como também de outros microrganismos presentes na amostra como fungos, arquea e protistas.
Além da identificação, o sequenciamento metagenômico permite a caracterização e quantificação de genes funcionais, fornecendo informações sobre as funções dos microrganismos presentes na amostra.
A tecnologia utilizada no MyBiome também se destaca pela alta resolução em comparação com a análise convencional de amplificação do gene 16S rRNA. Enquanto o sequenciamento do gene 16S não permite discriminar entre espécies bacterianas com regiões 16S semelhantes ou idênticas (34), o sequenciamento metagenômico permite uma identificação mais precisa dos microrganismos e seus genes funcionais.
Essa limitação implica em perda de informações significativas, pois diferentes espécies de um mesmo gênero podem ter funções muito distintas (35). O sequenciamento massivo de todo o genoma utilizado no MyBiome é atualmente a técnica de maior resolução para identificar microrganismos e detectar seus genes funcionais.
Essas características, aliadas a recomendações personalizadas, tornam o MyBiome uma análise única no mercado, proporcionando insights detalhados sobre a microbiota intestinal e sua funcionalidade.
O MyBiome é especialmente indicado para:
A realização de exames precisos e atualizados, como o MyBiome, é essencial para a realização de diagnósticos mais assertivos e para o melhor direcionamento dos tratamentos. E a SYNLAB está aqui para te ajudar.
Oferecemos soluções diagnósticas com rigoroso controle de qualidade às empresas, pacientes e médicos que atendemos. Estamos no Brasil há mais de 10 anos, atuamos em 36 países e três continentes, e somos líderes na prestação de serviços na Europa.
Entre em contato com a equipe SYNLAB e conheça os exames disponíveis.
Referências Bibliográficas
(1) Berg G, et al. Microbiome definition re-visited: old concepts and new challenges. Microbiome. 2020 Jun 30;8(1):103. doi: 10.1186/s40168-020-00875-0.
(2) Human Microbiome Project Consortium. Structure, function and diversity of the healthy human microbiome. Nature. 2012;486(7402), 207-214.
(3) Sun J; Kato I. Gut microbiota, inflammation and colorectal câncer. Genes Dis. 2016 Jun;3(2):130-143.
(4) Schoeler Marc; Caesar Robert. Dietary lipids, gut microbiota and lipid metabolismo. Reviews in Endocrine and Metabolic Disorders (2019) 20:461–472.
(5) Atanu Adak; Mojibur R. Khan. An insight into gut microbiota and its functionalities. Cellular and Molecular Life Sciences (2019) 76:473–493.
(6) Sonnenburg JL, Backhed F. Diet-microbiota interactions as moderators of human metabolism. Nature. 2016;535(7610):56–64.
(7) Sarkar A, Lehto SM, Harty S, Dinan TG, Cryan JF, Burnet PW. (2016) Psychobiotics and the manipulation of bacteria–gut–brain signals. Trends Neurosci 39(11):763–781.
(8) LeBlanc, J.G. et al. (2013) Bacteria as vitamin suppliers to their host: a gut microbiota perspective. Curr. Opin.Biotechnol. 24, 160–168
(9) Claus, S.P. et al. (2016) The gut microbiota: a major player in the toxicity of environmental pollutants? NPJ Biofilms Microbiomes 2, 16003
(10) Kamada, N. et al. (2013) Role of the gut microbiota inimmunity and inflammatory disease. Nat. Rev.Immunol. 13, 321–335).
(11) Ursell LK, Metcalf JL, Parfrey LW, Knight R. Defning the human microbiome. Nutr Ver. 2012;70(Suppl 1):S38-44.
(12) Hollister, E. B. et al. Structure and function of the healthy pre-adolescent pediatric gut microbiome. Microbiome. 2015;3,36.
(13) Claesson, M. J. et al. Gut microbiota composition correlates with diet and health in the elderly. Nature. 2012;488,178–184.
(14) Stewart, C. J. et al. Temporal development of the gut microbiome in early childhood from the TEDDY study. Nature. 2018;562,583–588.
(15) Arrieta MC, Stiemsma LT, Amenyogbe N, Brown EM, Finlay B. The intestinal microbiome in early life: health and disease. Front. Immunol. 2014;5,427.
(16) Le Chatelier, E. et al. Richness of human gut microbiome correlates with metabolic markers. Nature. 2013;500,541–546.
(17) Cryan, J. F., O’Riordan, K. J., Sandhu, K., Peterson, V. & Dinan, T. G. The gut microbiome in neurological disorders. Lancet Neurol. 2020;19,179–194.
(18) Nejman, D. et al. The human tumor microbiome is composed of tumor type specific intracellular bacteria. Science. 2020;368,973–980.
(19) Hepworth, M. R. et al. Innate lymphoid cells regulate CD4+ T-cell responses to intestinal commensal bacteria. Nature. 2013;498, 113–117.
(20) Rackaityte E, Lynch SV. The human microbiome in the 21st century. Nat Commun. 2020 Oct 16;11(1):5256. doi: 10.1038/s41467-020-18983-8.
(21) Scharschmidt, T. C. et al. A wave of regulatory T Cells into neonatal skin mediates tolerance to commensal microbes. Immunity. 2015;43,1011–1021.
(22) Qing He, Yuan Gao, Zhuye Jie, Xinlei Yu, Janne Marie Laursen. Two distinct metacommunities characterize the gut microbiota in Crohn’s disease patients. Gigascience. 2017 Jul 1;6(7):1-11.
(23) Katsuyoshi Matsuoka 1, Takanori Kanai. The gut microbiota and inflammatory bowel disease. Semin Immunopathol. 2015 Jan;37(1):47-55.
(24) He Q, et al. Two distinct metacommunities characterize the intestinal microbiota in patients with Crohn’s disease. Gigascience. 2017 Jul 1;6(7):1-11.
(25) Marchesi JR, Dutilh BE, Hall N, et al. Towards the human colorectal cancer microbiome. PLoS One. 2011;6:e20447.
(26) Borges-Canha M, et al. Role of colonic microbiota in colorectal carcinogenesis: a systematic review. Rev Esp Enferm Dig. 2015;107.
(27) Constante M, et al. Biogeographic Variation and functional pathways of the Gut microbiota in Celiac Disease. Gastroenterology 2022 163(5):1351-1363.e15.
(28) Radjabzadeh D, eta l. Gut microbiome-wide association study of depressive symptoms. Nat Commun. 2022. 6;13(1)7128.
(29) Cresci G, Bawden E. The Gut Microbiome: What we do and don’t know. Nutr Clin Pract. 2015 December; 30(6): 734–746.
(30) Ranjan R, Rani A, Metwally A, McGee HS, Perkins DL. Analysis of the microbiome: Advantages of whole genome shotgun versus 16S amplicon sequencing. Biochem Biophys Res Commun. 2016;22;469(4):967-77.
(31) Yang B, Wang Y, Qian PY. Sensitivity and correlation of hypervariable regions in 16S rRNA genes in phylogenetic analysis. BMC Bioinformatics. 2016 Mar 22;17:135.
(32) Větrovský T, Baldrian P. The variability of the 16S rRNA gene in bacterial genomes and its consequences for bacterial community analyses. PLoS One. 2013;8(2):e57923.
(33) Kumar S, Krishnani KK, Bhushan B, Brahmane MP. Metagenomics: Retrospect and Prospects in High Throughput Age. Biotechnol Res Int. 2015;2015:121735.
(34) Fox GE, Wisotzkey JD, Jurtshuk P Jr. How close is close: 16S rRNA sequence identity may not be sufficient to guarantee species identity. Int J Syst Bacteriol.1992 Jan;42(1):166-70.
(35) Clarridge JE 3rd. Impact of 16S rRNA gene sequence analysis for identification of bacteria on clinical microbiology and infectious diseases. Clin Microbiol Rev. 2004 Oct;17(4):840-62.
Conheça o exame
Doenças hepáticas representam uma carga significativa para a saúde global, com…
Continuar lendo
A doença de Alzheimer representa um dos maiores desafios globais em saúde…
Continuar lendo
A nutrigenética e a nutrigenômica têm se consolidado como áreas…
Continuar lendo